|
Sting
Millennium Suite: ferramentas para análise estrutural de
proteínas
Paula
Kuser Falcão, Michel Yamagishi,
Roberto Higa e Goran Neshich
O uso
da bioinformática é imprescindível para atender
aos desafios científicos oriundos do gigantesco volume de
dados produzidos pelos atuais projetos na área biológica.
Nesta era pós-genômica, o desafio é determinar
a função e o papel biológico de cada uma das
seqüências de proteínas que foram determinadas. Se
os genes são os portadores das instruções que
permitem a "construção" de um determinado
organismo, as proteínas são as responsáveis
por sua estrutura e funcionamento. No homem, são essas moléculas
que formam os ossos, músculos e os demais tecidos e comandam
o metabolismo. Defeitos ou malfuncionamento delas podem causar várias
doenças.
Um
organismo vivo contém em torno de 50.000 diferentes tipos
de proteínas. Cada proteína deve assumir sua conformação
específica para ser capaz de exercer sua função
biológica corretamente. Quando essa conformação
não é atingida, a proteína assume uma estrutura
que pode causar seu malfuncionamento. Algumas doenças como
anemia falciforme, que ocorre quando há uma mudança
de um aminoácido na proteína beta-globina; fibrose
cística, onde a proteína reguladora de condutância
transmembrana sofre uma alteração causando um transporte
anormal de íons de cloro; a catarata, causada por uma alteração
na proteína gama-cristalina; fenilcetonúria, um erro
no metabolismo que causa retardo mental devido ao mau funcionamento
da enzima fenilanina hidroxilase, são exemplos de doenças
que estão relacionadas com o mal funcionamento de proteínas.
Como
a função das proteínas está ligada à
sua estrutura, o conhecimento detalhado desta estrutura ajuda a
entender o papel de cada proteína. Mesmo depois de desvendar
a estrutura das proteínas, é preciso entender suas
interações, classificar as proteínas, encontrar
relações entre elas, identificar padrões, estudar
seu funcionamento. Quanto mais conhecermos sobre as proteínas
e suas estruturas, mais dados teremos para saber como ocorrem determinadas
doenças e até desenhar novas moléculas para
medicamentos.
Sabendo
que a informação bem organizada sobre seqüência,
estrutura e função de proteínas é uma
plataforma importante para entender os processos que ocorrem nos
seres vivos, assim como para o desenho de novas substâncias,
desenvolvemos um conjunto de ferramentas chamado STING
Millennium Suite (Figura 1). A característica principal
do STING Millennium é sua habilidade de combinar o fornecimento
de dados através da web com ferramentas de análise
estrutural, fazendo do SMS um instrumento completo para estudos
de macromoléculas.
| clique
na imagem para ampliar |
 |
| Página
inicial do software SMS indicando a atual versão do
softwaret SMS, a cidade e país do servidor e três
opções de acesso: a) páginas ilustradas
com imagens artísticas; b) páginas ilustradas
com ilustrações de moléculas; c) páginas
para usuários que têm monitores de baixa resolução
e conexão lenta com a internet. |
Ferramentas
para visualização e análise de estruturas
de proteínas
A
visualização de dados é uma área de
especial interesse no campo da bioinformática onde é
feito um grande esforço para apresentar parâmetros
medidos e calculados através do uso combinado de sistemas
de coordenadas, números, palavras, linhas, pontos, quadrantes
e cores. A representação gráfica é
quase sempre a maneira mais efetiva de descrever, explorar e condensar
um grande volume de informações numéricas,
tornando os gráficos em instrumentos poderosos para a análise
da informação quantitativa.
Juntos,
os dados de seqüência, características físico-química,
estrutura e função das proteínas, entre outros,
fornecem uma multiplicidade de informações cruciais
para entender os processos biológicos. Existem, atualmente,
quase 22.000 estruturas de proteínas disponíveis no
banco de dados Protein Data Bank (PDB)
e este número está aumentando a cada ano. Uma maneira
simples e rápida de acessar toda a informação
contida nos bancos de dados de proteínas é fundamental
para trabalhar com esse enorme volume de dados estruturais.
STING
Millennium (SMS) é uma suíte de programas com ferramentas
para a visualização e análise estrutural de
proteínas. Estes programas (módulos) estão
concentrados em um único pacote que visa oferecer um instrumento
completo para estudos das macromoléculas, suas estruturas
e as relações estrutura-função. Informações
como posição dos aminoácidos na seqüência
e na estrutura, busca de padrões, identificação
de vizinhança, ligações de hidrogênio,
ângulos e distâncias entre átomos, são
facilmente obtidas. Além disso, dados sobre natureza e volume
dos contatos atômicos inter e intra-cadeias, a conservação
e relação entre os contatos intra-cadeia, e parâmetros
funcionais, são questões que o usuário pode
responder sobre sua proteína utilizando o SMS.
O STING
Millennium, uma abreviatura de Sequence To and
withIN Graphics, utiliza tanto bancos de dados
públicos (PDB, HSSP, Prosite) como bancos de dados construídos
localmente (contatos, contatos de interface, acessibilidade, alinhamento
múltiplo de seqüências, regiões potenciais
para interação com fármacos, cavidades, potencial
eletrostático na superfície). Todo esse pacote de
análise foi implementado em uma interface amigável
ao usuário que pode ser acessada via web. Com a utilização
do SMS é possível analisar a relação
seqüência-estrutura-função, a qualidade
da estrutura, a natureza e o volume dos contatos atômicos
das cadeias intra e intermoleculares, a conservação
relativa dos aminoácidos em posições específicas
da seqüência baseados no alinhamento múltiplo
das seqüências, indicar aminoácidos que podem
ser essencias para a conformação da proteína
baseando-se na relação da conservação
dos resíduos envolvidos em contatos intra-cadeias, analisar
a geometria das distâncias Ca - Ca and Cß - Cß,
etc.
Os
módulos do SMS fornecem uma combinação de ferramentas
que permitem realizar uma excelente análise de uma determinada
estrutura de proteína.
| clique
na imagem para ampliar |
 |
| Exemplo
de funcionamento do pacote SMS e alguns módulos: (A)
janela da seqüência, de onde todos os módulos
do SMS podem ser acessados. A seqüência é
colorida de acordo com as características físico-químicas
dos aminoácidos. A medida que o usuário passa
o mouse sobre a seqüência, as informações
vão aparecendo na caixa "residue info". Dois
menus são também mostrados, demonstrando as escolhas
do SMS. (B) Módulo Protein Dossier - fornece um sumário
gráfico de várias características estruturais
importantes de uma proteína. Protein Dossier mostra um
cartoon da seqüência de aminoácidos acompanhados
de vários parâmetros anotados com escalas de cores,
representando cada aminoácido. O significado das cores
estão na parte de cima da imagem. Os contatos internos
e da interface da proteínas são mostrados acima
da seqüência dos aminoácidos, coloridos de
acordo com o tipo do contato. As outras linhas indicam vários
outros parâmetros do aminoácidos da proteína,
coloridos de acordo com seus valores e características,
permitindo que em uma única olhada no dossier da proteína
o usuário já consiga definir várias características
da proteína em estudo. (C) representação
tri-dimensional da estrutura em estudo. As duas superfícies
representam a interface entre as cadeias E (branca) e cadeia
I (rosa) da proteína. |
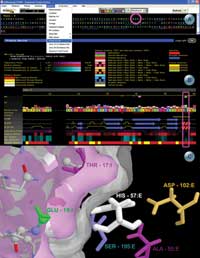
| clique
na imagem para ampliar |
 |
| Exemplo
de alguns módulos do pacote SMS: (A) diagrama Java das
distâncias entre os átomos carbono beta; (B) diagrama
ConSSeq apresentando a seqüência de aminoácidos
da proteína colorida de acordo com a conservação,
barras com códigos de cores representando a entropia
relativa, e informações sobre os aminoácidos
presentes em outras seqüências homólogas,
com sua respectiva freqüência; (C) tabela com a listagem
dos contatos atômicos mostrando o nome do aminoácido,
seu número, seu par no contato, o tipo de contato que
ocorre, a distância entre átomos que estão
fazendo contato e acessibilidade e entropia de dois aminoácidos
que estão em contato; (D e E) representação
gráfica dos contatos atômicos entre os aminoácidos
da proteína; (F) representação gráfica
do diagrama de Ramachandran utilizando todas as vantagens da
linguagem de programação Java. Este programa permite
uma interconexão dos dados dos ângulos diedrais
com a estrutura tri-dimensional da molécula; (G) cálculo
da freqüência dos aminoácidos que estão
na vizinhança tridimensional. |
Nas
figuras 2 e 3 mostramos uma colagem de vários módulos
do SMS durante a sessão de análise da proteína
alfa-quimotripsina (cadeia E) em complexo com uma cadeia de aminoácidos
denominada ovomucóide (cadeia I) da estrutura encontrada
no banco de dados PDB com código 1cho.pdb.
O STING
Millennium é composto de duas janelas principais. A "janela
da seqüência" (Fig. 2,A) mostra a seqüência
de aminoácidos da proteína e um menu com vários
comandos, e a "janela da estrutura" que mostra a estrutura
tri-dimensional da proteína. (Fig. 2C)
Cada
aminoácidos da janela da seqüência pode ser "clicado"
resultando na apresentação da sua posição
na estrutura tri-dimensional da proteína na janela da estrutura.
Nosso
mais recente produto, o Java Protein Dossier (JPD) é um banco
de dados e ferramenta de visualização que leva em
conta o novo conceito de comunicar muita informação
através de um único gráfico. O JPD fornece
aos usuários uma vasta coleção de parâmetros
físico-químicos descrevendo a estrutura da proteína,
estabilidade, função e interações com
outras macromoléculas. JPD utiliza a tecnologia Java com
um nível excepcional de interatividade cruzando referências
com a estrutura da proteína. Ao mesmo tempo o JPD é
um passo na direção de compilar uma base de dados
diversificada de descritores de estrutura-função que
podem ser usados como uma plataforma para a aquisição
de novos conhecimentos. JPD pode mostrar e analisar, simultaneamente,
todos os parâmetros físico-químicos de estruturas
que tenham sido previamente superimpostas, permitindo uma comparação
direta de parâmetros entre estruturas similares.
SMS como ferramenta didática
Essas
ferramentas de análise estrutural de proteínas são
desenvolvidas no Grupo de Bioinformática Estrutural da Embrapa/CNPTIA,
localizado dentro do campus da UNICAMP em Campinas, SP, Brasil.
Nossas
bases de dados são o resultado da análise computacional
dos dados disponíveis em fontes públicas e são
desenvolvidas para ajudar a entender a relação entre
as seqüências, estruturas e funções da
proteínas.
O software
SMS está sendo espelhado desde 2001 em San Diego, no site
do Protein Data Bank (PDB), o mais completo banco de dados de estruturas
de proteínas. Além deste espelho, temos também
versões do SMS espelhados na Universidade de Columbia em
Nova Iorque; em Madrid, na Espanha; e em La Plata, na Argentina.
O Grupo
de Bioinformática Estrutural desenvolve projetos em colaboração
com alguns grupos de pesquisa no Brasil e no mundo, com o objetivo
de aumentar o espectro de informações oferecidas através
de nossos produtos e também para aplicar as ferramentas desenvolvidas
adquirindo conhecimento em sistemas biológicos específicos.
O laboratório
tem uma infraestrutura que permite a implementação
de projetos de pesquisa que necessitam de uma quantidade significativa
de CPU, com equipamentos computacionais de última geração,
assim como uma infraestrutura que nos permite organizar cursos de
bioinformática estrutural para estudantes.
STING
Millennium Suite está descrito aqui como um pacote que engloba
várias ferramentas de análise de estruturas de proteínas
num único endereço. O usuário pode fazer o
download e instalação do pacote completo do SMS em
sua própria plataforma.
A partir
do SMS também é possível acessar um grande
número de links para outros bancos de dados de proteínas
e também outros programas. Este extenso cruzamento de dados
permite que o usuário tenha um ambiente completo e integrado
para a análise seqüência/estrutura/função
de uma proteína.
Paula
Kuser Falcão, Michel Yamagishi, Roberto Higa e Goran Neshich
são pesquisadores do Centro de Bioinformática Estrutural
da Embrapa Informática Agropecuária
|